MRMメソッドの作成
ターゲットタンパクを感度よく定量するためのMRMメソッドを作成します。ここでは、高速液体クロマトグラフ―三連四重極質量分析装置(LC-MS)の使用を前提に説明します。例として島津製作所製nanoLCとLCMS8060を使用する場合を説明します。Skylineは、島津製作所製LCMS80x0シリーズに対応済です(後述)。
MRMメソッドを作成するには、ターゲットタンパクのアミノ酸配列から
- LC-MSで感度よく検出できるトリプシン消化ペプチドを選抜
- 三連四重極質量分析装置(トリプルQ)で感度よく検出できるMRM系列を設定する必要があります。
注:コリジョン電圧等のフラグメンテーションのパラメーターはプリカーサーイオンのm/zに依存して設定します。Skylineが島津製作所製LCMS80x0シリーズに最適な値を自動的に設定してくれます。
ヒトであれば、すでに全タンパクについてMRMメソッドが作成済みですのでそれを使いましょう。一方、マニアックな生物でマニアックなタンパクが定量したい場合はMRMメソッドを自力で作成する必要があります。
例えば大腸菌 E. coli のPgkタンパクを測定するMRMメソッドを作成したいとします。
- 大腸菌 E. coli のPgkタンパクを含有している粗タンパク液をゲットする(精製したPgkタンパクとか、Pgkタンパクを過剰発現した大腸菌遺伝子組換え株の粗酵素液とか、Pgkタンパクが発現しているはずの大腸菌野生株の粗酵素液などを作成します)。
- トリプシン消化した、ペプチドサンプルを調製する。
- Pgkタンパクのアミノ酸配列から、すべての候補ペプチドを生成し、それらの全候補MRM系列を生成し、「MRMメソッド作成用のMRMメソッド」を生成する。
- 「MRMメソッド作成用のMRMメソッド」で「ペプチドサンプル」サンプルを分析する。
- 高強度で大腸菌のPgkタンパクを定量できるペプチドとMRM系列を選抜する。
という作業を行います。このうち3と5がSkylineの出番です。
FASTA形式のアミノ酸配列をコピペすればいい
まず、大腸菌 E. coli のPgkタンパクのアミノ酸配列のデータをFASTA形式でゲットしましょう。
http://www.uniprot.org/uniprot/P0A799
>sp|P0A799|PGK_ECOLI Phosphoglycerate kinase OS=Escherichia coli (strain K12) GN=pgk PE=1 SV=2
MSVIKMTDLDLAGKRVFIRADLNVPVKDGKVTSDARIRASLPTIELALKQGAKVMVTSHL
GRPTEGEYNEEFSLLPVVNYLKDKLSNPVRLVKDYLDGVDVAEGELVVLENVRFNKGEKK
DDETLSKKYAALCDVFVMDAFGTAHRAQASTHGIGKFADVACAGPLLAAELDALGKALKE
PARPMVAIVGGSKVSTKLTVLDSLSKIADQLIVGGGIANTFIAAQGHDVGKSLYEADLVD
EAKRLLTTCNIPVPSDVRVATEFSETAPATLKSVNDVKADEQILDIGDASAQELAEILKN
AKTILWNGPVGVFEFPNFRKGTEIVANAIADSEAFSIAGGGDTLAAIDLFGIADKISYIS
TGGGAFLEFVEGKVLPAVAMLEERAKK
FASTA形式のアミノ酸配列のデータをコピーし、Skylineの「ターゲット」領域内で「右クリック」=>「貼り付け」すると候補ペプチドのリストが生成します。
各ペプチドはK.MTDLDLAGK.R [5, 13]のように記載さていますが、これは5-13アミノ酸残基のペプチドMTDLDLAGKでN末端側の1つ隣の残基はKで、C末端側の1つ隣の残基はRという意味です。出てくるペプチドのリストは設定によって異なります。
また、右下に現在のタンパク数、ペプチド数、プリカーサー数、MRM系列数が表示されています。
まずはこれを読むべし
定量プロテオーム解析を志す方必携は下記論文です。プロテオーム解析の基本的な考え方、メソッド作成法、サンプル調製法の詳細が事細かに解説されています。プロテオーム解析をうまくやるコツは、まずはここに書いてある通りにやってみること。といっても過言ではありません。Open Access論文にしてくださって感謝であります。
Uchida, Y., Tachikawa, M., Obuchi, W., Hoshi, Y., Tomioka, Y., Ohtsuki, S., and Terasaki, T. (2013). A study protocol for quantitative targeted absolute proteomics (QTAP) by LC-MS/MS: application for inter-strain differences in protein expression levels of transporters, receptors, claudin-5, and marker proteins at the blood-brain barrier in ddY, FVB, and C57BL/6J mice. Fluids Barriers CNS 10, 21.
ペプチド設定
候補ペプチドを選ぶ基準を設定するのが、メニュー「設定」=>「ペプチド設定」です。
タブがたくさんありますが、このうち「消化」「フィルタ」タブが候補の選抜にかかわります。
酵素:通常はTrypsin[KR|P]でOKです。
最大ミス開裂数:許容するmiss cleavageの数を設定します。切れるはずのサイトで切れないmiss cleavageは定量プロテオミクスでは取り扱いが難しいので0にしておきましょう。
バックグラウンドプロテオーム:候補ペプチドが同生物種の他タンパクにも含まれていないかを確認するためのものです。動作がよくわからないので今回は使わずにおいておきます。
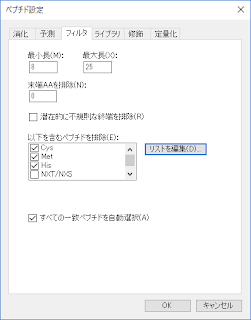
「フィルタ」タブでは下記を設定します
最小長:選択するペプチドの長さの最小です。この場合8残基以上のペプチドを利用します。
最大長:選択するペプチドの長さの最大です。この場合8残基以上のペプチドを利用します。
ターゲットプロテオーム解析ではおおよそ8-25残基程度のペプチドを選抜することが普通です。これより短いと特異性が下がり、これより長いとイオン化効率、クロマト上での挙動が悪くなることが増えるようです。一方、ペプチドの候補が少ないときは、この幅を広げて少しでも候補を増やしたりもします。
末端AAを排除:この長さだけ5’および3’末端領域を無視するという設定です。端っこは定量には向かないらしいのですが、いつも0にしています。
潜在的に不規則な終端を排除:よくわからないのでチェックなし
以下を含むペプチドを排除:Cys, Met, Hisなどを含むペプチドは酸化、イオン化効率が低いなどの理由で候補ペプチドに向かないといわれていますが。調べたところ、Cys, Met, Hisなどを含むペプチドもわりと普通にターゲットペプチドで使われているようですので、まずはフィルターをかけずに選抜し、候補が多すぎるときにはフィルターをかけて候補を減らす。というような使い方をしています。
すべての一致ペプチドを自動選抜:チェックと入れています。
このようにしてフィルターをペプチド設定を行うと下記のようになり、「ターゲット」のペプチド数が減っていました。



0 件のコメント:
コメントを投稿