定量プロテオーム解析用のメソッド作成
Skylineは定量プロテオーム解析用のソフトウェアです。複数のタンパク質用のMRMメソッドを並列したメソッドを作成できます。今回はテキストファイルから定量プロテオーム解析用メソッド作成法を紹介します。
1.まずSkylineを起動し、「空のドキュメント」を開きます。
2.次に下記のようなタブ区切りのテキストファイルを用意します。こういうデータは論文のサプリメントデータとかについています。
タンパク名、ペプチド、Q1
m/
z, Q3
m/
zの順番です(どうもこの順番は適当でもSkylineがよきにはからってくれるようだ)。
serB
LAGSLTAR
394.735425944185
347.20429299249
serB
LAGSLTAR
394.735425944185
460.28835696962
serB
LAGSLTAR
394.735425944185
675.37896287917
serB
LAGSLTAR
394.735425944185
604.34184909446
hisB
FGFVLPMDECLAR
777.876353174435
1104.51817585726
hisB
FGFVLPMDECLAR
777.876353174435
519.27132700649
hisB
FGFVLPMDECLAR
777.876353174435
991.43411188013
hisB
FGFVLPMDECLAR
777.876353174435
1203.58658977025
hisB
LAFEPGVIPELLK
713.42177643763
965.60354308039
hisB
LAFEPGVIPELLK
713.42177643763
1094.64613616836
hisB
LAFEPGVIPELLK
713.42177643763
868.55077923154
hisB
LAFEPGVIPELLK
713.42177643763
1241.71455008135
hisB
VEGDTLPSSK
516.764577305065
804.4103225771
hisB
VEGDTLPSSK
516.764577305065
418.23017338716
hisB
VEGDTLPSSK
516.764577305065
632.3619158327
hisB
VEGDTLPSSK
516.764577305065
531.31423736429
pgk
SLYEADLVDEAK
676.833188179465
989.47913041291
pgk
SLYEADLVDEAK
676.833188179465
789.39942354023
pgk
SLYEADLVDEAK
676.833188179465
860.43653732494
pgk
SLYEADLVDEAK
676.833188179465
1152.54245894546
pgk
VLPAVAMLEER
614.34247202944
847.43476350313
pgk
VLPAVAMLEER
614.34247202944
748.36634959014
pgk
VLPAVAMLEER
614.34247202944
1015.52464113669
pgk
VLPAVAMLEER
614.34247202944
677.32923580543
3.テキストをクリップボードにコピーします。
4.左上青枠をクリックし、ペーストします(貼り付けて後複数個貼り付けても1つのメソッドで行ける)。
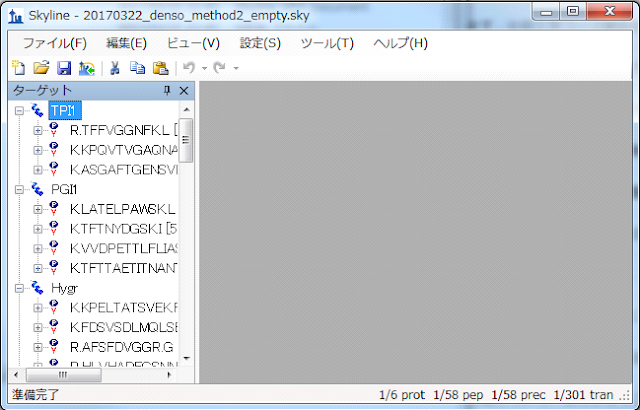
5.うまくいくと、ターゲット欄にタンパク質が登録されます。
6. 設定パラメータについては研究目的により異なるため、その都度入力します。
6-1.使用するペプチドの長さの範囲を指定する。
設定(S) →ペプチド設定→フィルタ・・・
設定例:
最小長(M):7 - 最大長(X):30、
末端AAを排除(N):25 ←今回は意味なし
□潜在的に不規則な終端を排除 ←空白のまま
以下を含むペプチドを排除
■Cys ←これらのアミノ酸が含まれるペプチドは定量に向いていないといわれています。
□Met
□His
■全ての一致ペプチドを自動選択(A)
6-2.修飾の設定
設定(S) →ペプチド設定→修飾・・・
■Carbamidomethyl(C)←還元アルキル化した場合はチェック
最大修飾(M)=3←よくわからない
最大ニュートラルロス(N)=1
同位体標識タイプ(T)=heavy ←安定同位体標識内部標準を用いる場合ここで設定
同位体修飾(I )
□label:15N 15N …フル15N標識の時
□label:13C 13C …フル13C標識の時
変更時は別記事の「Skyline_15N→13C_ペプチド設定_変更の方法」へ
内部標準タイプ(N)=heavy ←安定同位体標識内部標準を用いる場合heavyにしておく。
6-3.トランジションの設定
設定(S) →トランジションの設定→フィルタタブ
プリカーサ電荷(P)=2 ←2価と3価の場合は"2,3"とする。
イオン電荷(I)=1
イオンタイプ(T)=y ←yとb系列のときは"y,b"とする。
開始点(F)=イオン1 ←Q3を選ぶ基準
終了点(O)=最後のイオン
特別なイオン
■N-terminal to Proline
N末がプロリンになるペプチドは無視
プリカーサm/z排除ウインドウ [ ] m/z ←空白のまま
■一致する全てトラジションを自動選択(A)
7.ファイル名を付け、保存する。←… 解析メソッド出来上がり
<例>KT0031_Normal3-empty